Les empreintes génétiques
- Fiche de cours
- Quiz et exercices
- Vidéos et podcasts
- Définir la notion de séquences microsatellites.
- Comprendre la notion de polymorphisme.
- Comprendre l’origine du polymorphisme inter-individu.
- Comprendre le principe de la technique RFLP.
- Comprendre l’intérêt de la PCR dans l’optimisation de l’obtention des empreintes génétiques.
- Savoir identifier les liens de parenté entre différents individus à partir des empreintes génétiques.
Dans le génome de n’importe quelle espèce, il existe des séquences caractérisées par un polymorphisme élevé appelées microsatellites. Celles-ci peuvent être utilisées pour distinguer génétiquement les individus entre eux mais aussi pour établir des liens de parenté.
Deux techniques sont possibles. L’une est basée sur l’utilisation d’enzymes de restriction (technique RFLP) qui fragmentent le génome en plusieurs portions dont les tailles vont varier en fonction de la longueur de la séquence microsatellite. L’autre est basée sur une PCR qui permet l’amplification spécifique des locii où sont localisées les séquences microsatellites.
- Connaître l‘origine de la variabilité de l’ADN et des génotypes.
- Comprendre les notions d’hétérozygotie et d’homozygotie.
Seulement 10 % du génome humain est codé en protéines. Cette fraction du génome est variable puisque l’on peut constater l’existence des allèles.
Le reste du génome est lui aussi variable. Notamment, on note la présence de séquences répétées en tandem de 2 à 5 nucléotides appelées microsatellites ou STR (Sequence Tandem Repeat).
Ces séquences sont très nombreuses et réparties sur l’ensemble du génome. Le séquençage du génome humain a permis d’en localiser un grand nombre. Ces séquences microsatellites sont hautement polymorphes. Elles se distinguent d’un individu à un autre par le nombre de répétitions. Ainsi, un individu possède pour chaque STR sa propre répétition qu’il aura hérité de ses parents. Aussi, il pourra exister une hétérozygotie pour les séquences microsatellites. En étudiant un grand nombre de STR chez un individu, on peut déterminer son empreinte génétique propre avec une infime probabilité pour qu’on la retrouve à l’identique chez un autre individu (excepté les vrais jumeaux). Par contre, cet individu présente certaines séquences communes avec ses parents et ses frères et sœurs ce qui permettra d’établir un lien de parenté.
C’est Alex Jeffreys qui en 1984 met en évidence le polymorphisme des microsatellites dans le génome humain. Il montre qu’il est possible de distinguer un individu des autres grâce à ces séquences mais aussi d’établir des liens de parenté entre les individus.
La mise au point de la technique de PCR par Kary Mullis (1986) ainsi que la connaissance de la séquence des génomes permet aujourd’hui d’optimiser la technique des empreintes génétiques.
Les locii d’un grand nombre de STR sont aujourd’hui connus. La PCR permet d’amplifier spécifiquement ces régions du génome et d’obtenir des fragments dont la taille va dépendre du nombre de répétitions dans la séquence microsatellite.
La PCR est réalisée à l’aide d’une amorce couplée à un fluorochrome. Les fragments d’ADN sont ensuite séparés par chromatographie. Chaque fragment est alors reconnu et analysé selon sa taille. Cette technique est aujourd’hui utilisée pour établir les tests de paternité et les filiations entre individus ainsi que par la police scientifique qui compare systématiquement 13 loci et 2 loci situés sur les chromosomes sexuels.
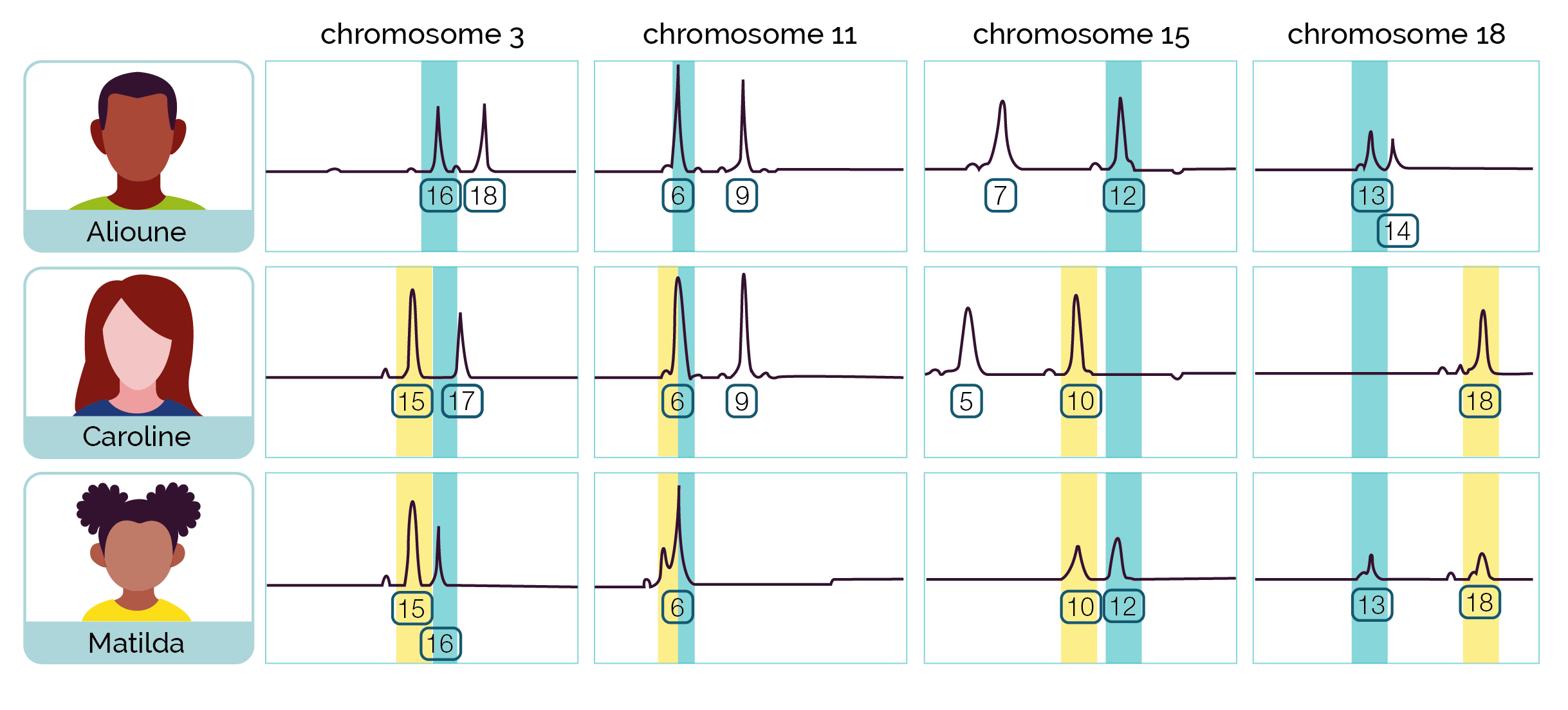
Exemple de test de paternité
Les STR sont présents dans tous les génomes des êtres vivants. Il est donc possible d’élargir cette technique à d’autres espèces aussi bien animales que végétales. Ceci devient aussi un outil performant pour la phylogénie moléculaire.
Une autre technique peut être utilisée basée sur la grande spécificité des enzymes de restriction pour une séquence nucléotidique restreinte à 3 à 5 nucléotides. La mutation d’un seul nucléotide de cette séquence consensus annule la capacité de l’enzyme de couper la double hélice d’ADN.
Pour réaliser son étude, il utilise des enzymes de restriction (technique RFLP-Restriction Fragment Lengh Polymorphism). Ce sont des enzymes bactériennes capables de reconnaître et de couper de courtes séquences consensus dans l’ADN. Il réalise alors la fragmentation de différents échantillons d’ADN avec la même enzyme et sépare ensuite les fragments par électrophorèse. Il constate alors que même si les individus de même lignée présentent des fragments communs, ils possèdent tous des fragments qui permettent de les distinguer les uns des autres.
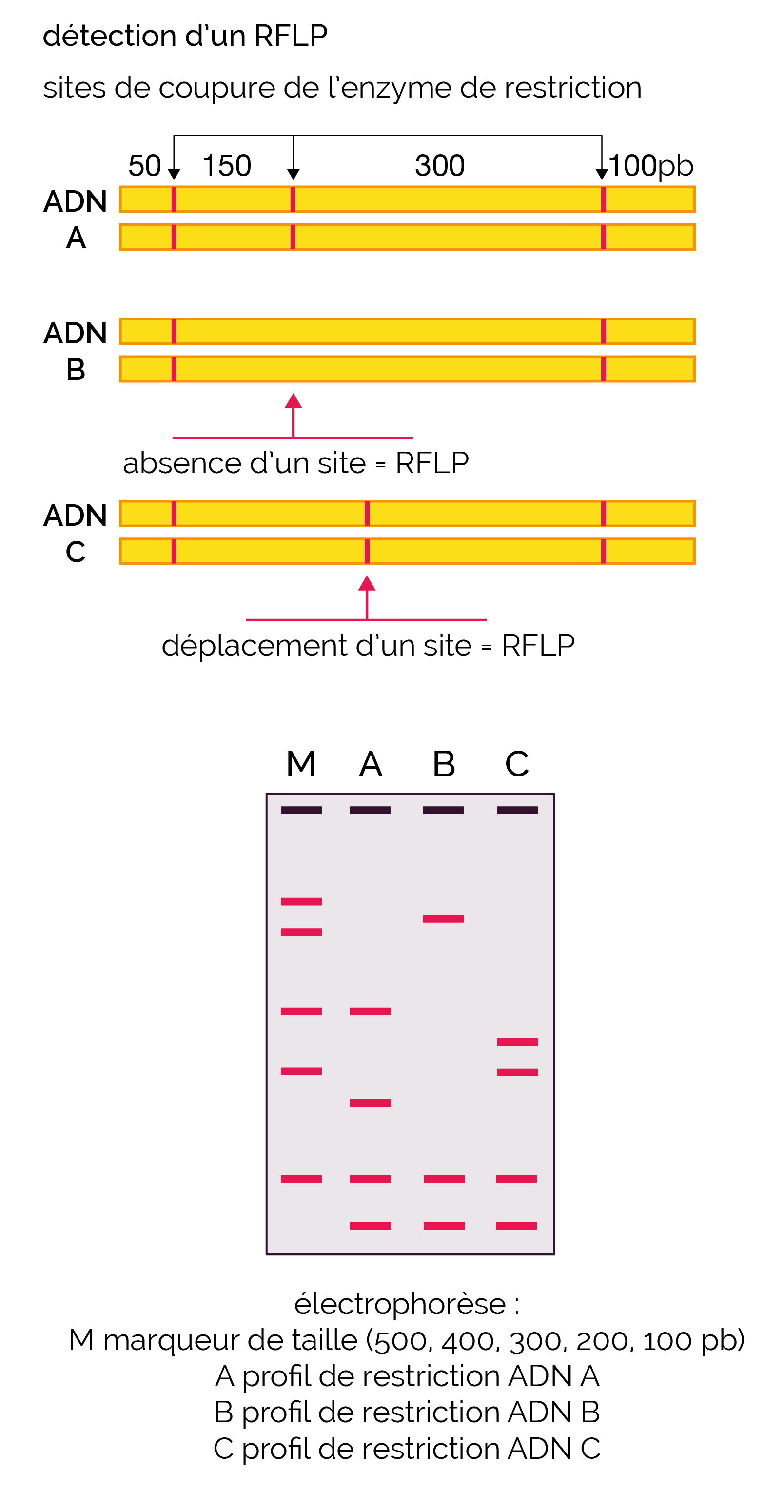
Détection d'un RFLP
.

Des quiz et exercices pour mieux assimiler sa leçon
La plateforme de soutien scolaire en ligne myMaxicours propose des quiz et exercices en accompagnement de chaque fiche de cours. Les exercices permettent de vérifier si la leçon est bien comprise ou s’il reste encore des notions à revoir.

Des exercices variés pour ne pas s’ennuyer
Les exercices se déclinent sous toutes leurs formes sur myMaxicours ! Selon la matière et la classe étudiées, retrouvez des dictées, des mots à relier ou encore des phrases à compléter, mais aussi des textes à trous et bien d’autres formats !
Dans les classes de primaire, l’accent est mis sur des exercices illustrés très ludiques pour motiver les plus jeunes.

Des quiz pour une évaluation en direct
Les quiz et exercices permettent d’avoir un retour immédiat sur la bonne compréhension du cours. Une fois toutes les réponses communiquées, le résultat s’affiche à l’écran et permet à l’élève de se situer immédiatement.
myMaxicours offre des solutions efficaces de révision grâce aux fiches de cours et aux exercices associés. L’élève se rassure pour le prochain examen en testant ses connaissances au préalable.

Des vidéos et des podcasts pour apprendre différemment
Certains élèves ont une mémoire visuelle quand d’autres ont plutôt une mémoire auditive. myMaxicours s’adapte à tous les enfants et adolescents pour leur proposer un apprentissage serein et efficace.
Découvrez de nombreuses vidéos et podcasts en complément des fiches de cours et des exercices pour une année scolaire au top !

Des podcasts pour les révisions
La plateforme de soutien scolaire en ligne myMaxicours propose des podcasts de révision pour toutes les classes à examen : troisième, première et terminale.
Les ados peuvent écouter les différents cours afin de mieux les mémoriser en préparation de leurs examens. Des fiches de cours de différentes matières sont disponibles en podcasts ainsi qu’une préparation au grand oral avec de nombreux conseils pratiques.
Des vidéos de cours pour comprendre en image
Des vidéos de cours illustrent les notions principales à retenir et complètent les fiches de cours. De quoi réviser sa prochaine évaluation ou son prochain examen en toute confiance !









