Le séquençage de l'ADN et la connaissance du génotype et du phénotype d'un individu
- Fiche de cours
- Quiz et exercices
- Vidéos et podcasts
- Connaitre une méthode de séquençage de l’ADN.
- Comprendre l’utilité des banques de données dans la découverte des gènes déterminant les phénotypes chez les organismes.
- La méthode de Sanger est une technique développée dans les années 1970 pour séquencer l’ADN.
- Le génome humain a été séquencé pour la première fois en 2003.
- Aujourd’hui, de nombreux génomes ont été séquencés. La bioinformatique permet de découvrir les différents gènes et allèles qui déterminent le phénotype.
- L’ADN est la molécule qui contient les gènes.
- Les gènes portent l’information génétique de l’organisme.
- Le phénotype dépend du génome et du génotype.
L’ADN est formé de deux brins qui sont des polymères de nucléotides possédant quatre bases azotées : adénine, guanine, cytosine, thymine. Séquencer une molécule d’ADN signifie « lire » la séquence de bases azotées qui composent chaque brin. Il est possible de séquencer tout type d’ADN (ou d’ARN), depuis des petits génomes de virus qui font quelques milliers de bases jusqu’à des génomes géants de plusieurs centaines de milliards de bases chez certains organismes.

Le séquençage du génome humain a été complété en 2003. Il a nécessité une collaboration internationale de milliers de chercheurs pendant plus d’une décennie, dans ce que l’on a appelé le Human Genome Project. Ceux-ci ont eu recours principalement à une méthode : le séquençage Sanger.
La méthode de Sanger est une des premières techniques pour séquencer l’ADN. Bien que considérée aujourd’hui comme assez lente et onéreuse, elle a été la principale technique utilisée lors du Human Genome Project et elle est encore régulièrement utilisée en laboratoire pour séquencer de petites molécules d’ADN, jusqu’à 900 kb (comme des plasmides bactériens).
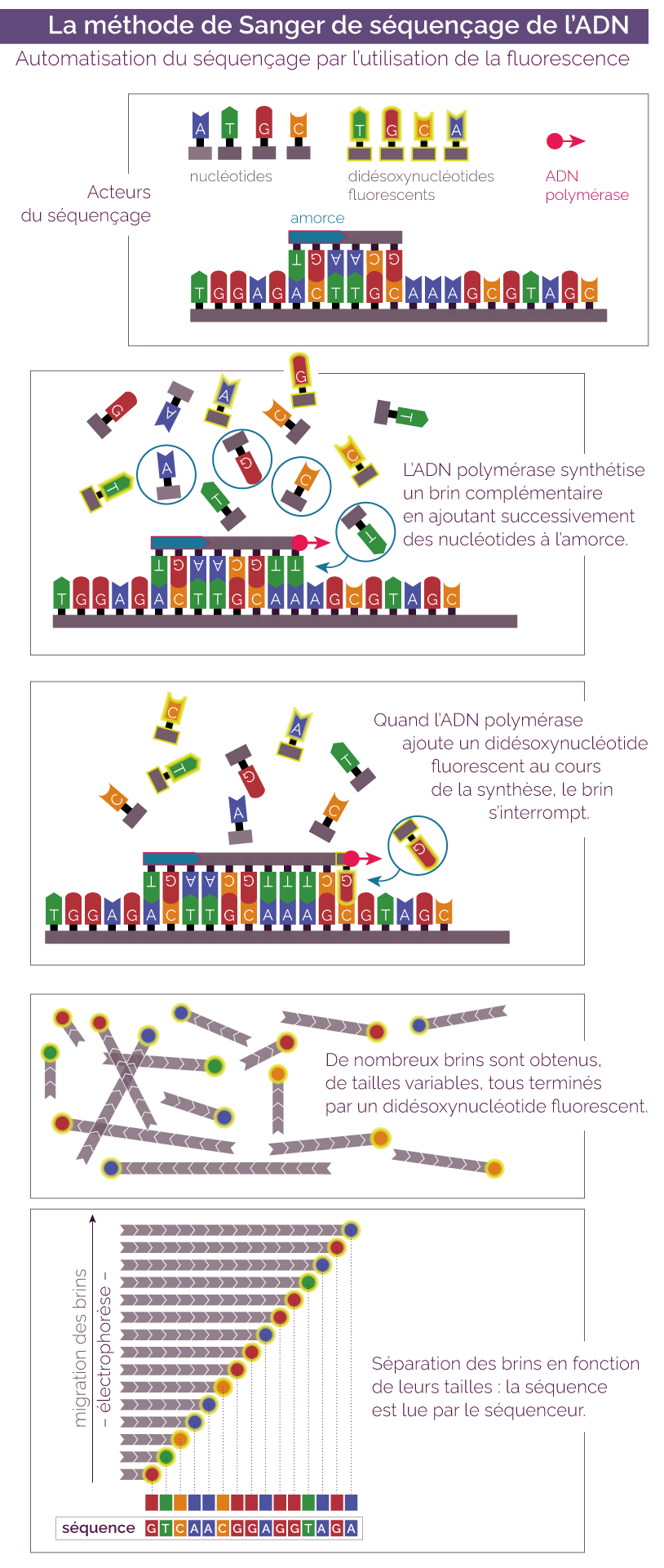
La méthode de Sanger, du nom de son créateur Frederick Sanger, nécessite tous les éléments utilisés pour une synthèse d’ADN, c’est-à-dire :
- une molécule d’ADN, qui est celle que l’on souhaite séquencer ;
- des ADN polymérases, qui sont des protéines capables de créer un brin complémentaire à partir d’un brin matrice ;
- des désoxyribonucléotides simples en grande quantité (nommés dNTP) avec les quatre bases azotées ;
- une amorce ARN pour démarrer la réaction de synthèse d’ADN. Elle doit posséder une séquence complémentaire de la région en aval de la séquence que l’on souhaite séquencer.
Il est également nécessaire d’y ajouter en petite quantité des nucléotides particuliers, qui n’existent pas dans la nature, les didésoxyribonucléotides (ddNTP). Ils sont légèrement différents des dNTP.
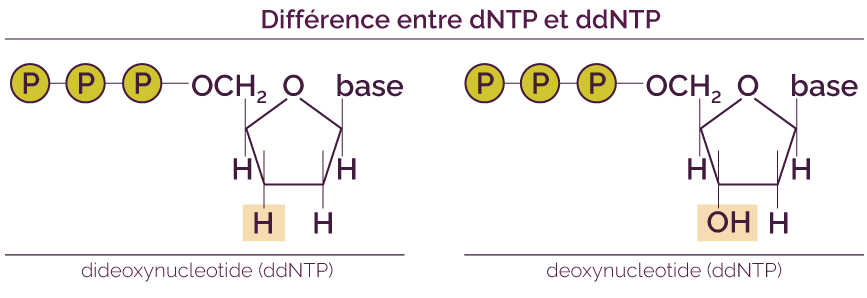
Il faut les quatre types de base ddNTP : ddATP, ddGTP, ddCTP et ddTTP. Chacun de ces ddNTP est marqué avec un pigment fluorescent (une couleur différente pour chaque base).
Les ddNTP sont intégrés au néobrin par l’ADN polymérase comme des désoxyribonucléotides normaux. Cependant, ce sont des terminateurs de réplication car il n’est pas possible d’ajouter un nucléotide au brin d’ADN après un ddNTP.
On place les réactifs ensemble dans un milieu de culture. La température est augmentée jusqu’à ce que les deux brins de l’ADN se séparent (à environ 75 °C). Cela permet la fixation de l’amorce d’ARN sur le brin matrice.
La température est ensuite abaissée, ce qui déclenche la réaction de synthèse de l’ADN. L’ADN polymérase produit un néobrin complémentaire au brin matrice en commençant au premier nucléotide après l’amorce d’ARN. Un par un les dNTP du milieu sont ajoutés au néobrin jusqu’à ce qu’un ddNTP soit intégré. Cela stoppe la synthèse du néobrin.
Enfin, on augmente à nouveau la température afin de séparer le néobrin du brin d’ADN matrice. On peut ainsi répéter l’opération un grand nombre de fois.
Quel est l’intérêt des ddNTP ?
Puisque les ddNTP sont en faible quantité, ils sont intégrés aléatoirement au brin synthétisé. En répétant l’opération un grand nombre de fois, on obtient des brins de toutes les longueurs possibles.
On soumet ensuite tous les brins obtenus à une électrophorèse sur gel de polyacrylamine. Cette technique sépare les brins d’ADN selon leur longueur, du plus petit au plus grand.
Enfin, chaque brin passe devant un laser qui détecte la couleur du pigment fluorescent de son ddNTP final. Ainsi, la séquence d’ADN à séquencer est reconstituée, nucléotide par nucléotide.
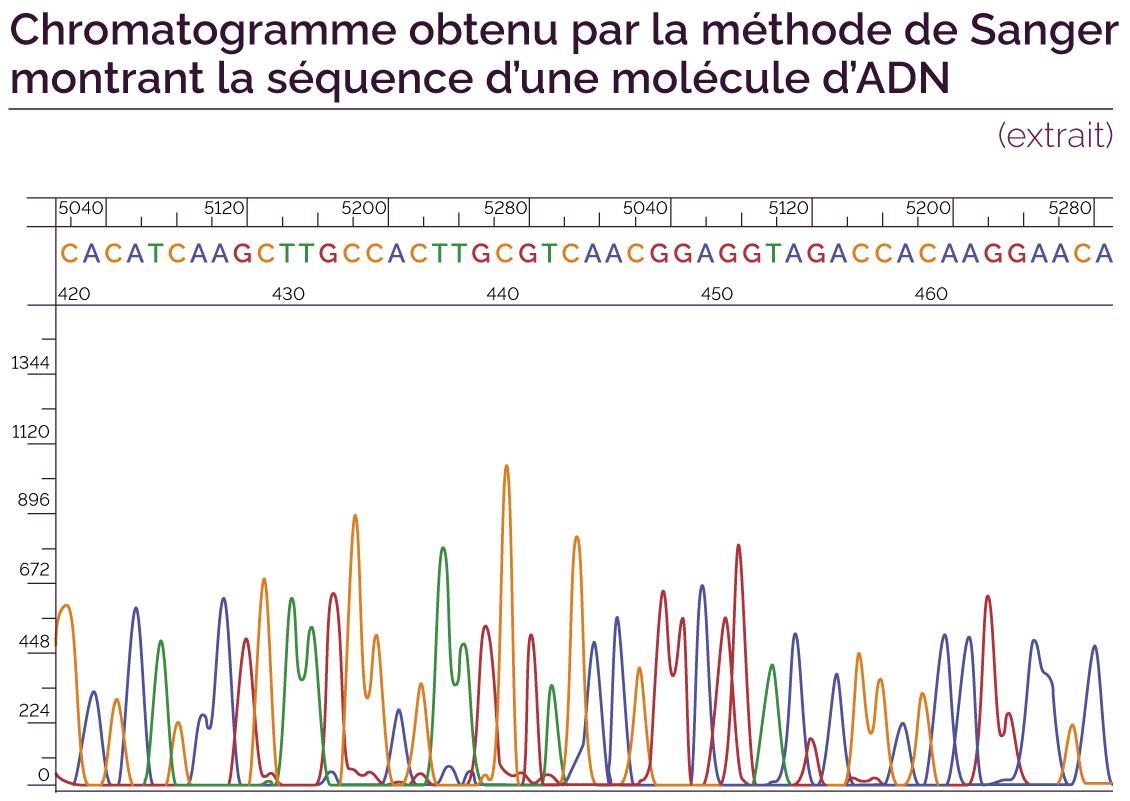
Voici un chromatogramme obtenu par la méthode de Sanger :
Depuis le début du XXIe siècle, les techniques de séquençage d’ADN se sont largement automatisées. Elles partagent cependant entre elles les mêmes caractéristiques :
- Elles fonctionnent en parallèle, c’est-à-dire que plusieurs séquences peuvent être lues en même temps ;
- Elles sont miniaturisées, c’est-à-dire que chaque réaction de séquençage peut être réalisée dans un espace très petit.
Ces améliorations techniques ont permis d’accélérer le séquençage de l’ADN et d’en diminuer le coût.
Ainsi le Human Genome Project a coûté des dizaines de millions de dollars, alors qu’aujourd’hui il est possible de séquencer le génome entier d’une personne en quelques jours pour environ 1000 dollars, soit environ 850 euros.
Avec trois milliards de paires de bases et environ 25 000 gènes, le génome humain n’est pas exceptionnellement grand. Il fait approximativement la même taille que celui de la souris, Mus musculus.
Néanmoins, l’analyse de ce génome est une tâche longue et difficile. Pour aider les chercheurs dans leur travail, des banques de données ont été mises en place sur Internet avec les nombreux génomes des différents individus séquencés jusqu’à présent.
La bioinformatique s’est énormément développée ces dernières décennies afin de créer des algorithmes capables d’analyser cette énorme quantité d’information.
En ayant accès à une large base de données de différents génomes, les scientifiques peuvent dès lors chercher dans le génome des variants (le plus souvent des allèles et des mutations spontanées) associés à un phénotype, par exemple, un risque plus grand de développer certaines maladies. Les GWAS (Genome Wide Association Studies, études d'associations pangénomiques) sont des études qui comparent un grand nombre de génomes. Elles permettent de découvrir les gènes et les allèles qui sont liés aux différents phénotypes.
Des études ont montré le lien entre plusieurs régions du génome et un risque accru de développer la mucoviscidose, en particulier le gène AGTR2 codant pour le récepteur à angiotensine de type 2.

Des quiz et exercices pour mieux assimiler sa leçon
La plateforme de soutien scolaire en ligne myMaxicours propose des quiz et exercices en accompagnement de chaque fiche de cours. Les exercices permettent de vérifier si la leçon est bien comprise ou s’il reste encore des notions à revoir.

Des exercices variés pour ne pas s’ennuyer
Les exercices se déclinent sous toutes leurs formes sur myMaxicours ! Selon la matière et la classe étudiées, retrouvez des dictées, des mots à relier ou encore des phrases à compléter, mais aussi des textes à trous et bien d’autres formats !
Dans les classes de primaire, l’accent est mis sur des exercices illustrés très ludiques pour motiver les plus jeunes.

Des quiz pour une évaluation en direct
Les quiz et exercices permettent d’avoir un retour immédiat sur la bonne compréhension du cours. Une fois toutes les réponses communiquées, le résultat s’affiche à l’écran et permet à l’élève de se situer immédiatement.
myMaxicours offre des solutions efficaces de révision grâce aux fiches de cours et aux exercices associés. L’élève se rassure pour le prochain examen en testant ses connaissances au préalable.

Des vidéos et des podcasts pour apprendre différemment
Certains élèves ont une mémoire visuelle quand d’autres ont plutôt une mémoire auditive. myMaxicours s’adapte à tous les enfants et adolescents pour leur proposer un apprentissage serein et efficace.
Découvrez de nombreuses vidéos et podcasts en complément des fiches de cours et des exercices pour une année scolaire au top !

Des podcasts pour les révisions
La plateforme de soutien scolaire en ligne myMaxicours propose des podcasts de révision pour toutes les classes à examen : troisième, première et terminale.
Les ados peuvent écouter les différents cours afin de mieux les mémoriser en préparation de leurs examens. Des fiches de cours de différentes matières sont disponibles en podcasts ainsi qu’une préparation au grand oral avec de nombreux conseils pratiques.
Des vidéos de cours pour comprendre en image
Des vidéos de cours illustrent les notions principales à retenir et complètent les fiches de cours. De quoi réviser sa prochaine évaluation ou son prochain examen en toute confiance !









